Genom ludzki: budowa, funkcje, HGP i projekt ENCODE
Genom ludzki: budowa i funkcje, odkrycia projektu HGP i najnowsze wnioski ENCODE o kontroli genów — przegląd sekwencji, zastosowań klinicznych i badań molekularnych.
Ludzki genom zapisany jest na 23 parach chromosomów w jądrze komórkowym oraz w małym mitochondrialnym DNA. Wiele wiadomo już o sekwencjach DNA, które znajdują się na naszych chromosomach. Częściowo wiadomo też, co tak naprawdę robi DNA. Zastosowanie tej wiedzy w praktyce dopiero się zaczyna.
Budowa i podstawowe informacje
Genom człowieka to kompletna informacja genetyczna zapisane w postaci sekwencji nukleotydów (A, T, C, G). W przybliżeniu haploidalny genom ludzkiego dorosłego człowieka zawiera około 3–3,2 miliarda par zasad. Chromosomy występują w komórkach somatycznych w postaci 23 par: 22 par autosomów oraz para płciowa (XX u kobiet, XY u mężczyzn). Dodatkowo komórki zawierają mitochondrialne DNA — małą, kolistą cząsteczkę (~16,6 kb), która koduje kilka niezbędnych białek łańcucha oddechowego oraz RNA.
- Około 20–25 tysięcy genów koduje białka; są to tzw. geny kodujące białka. Jednak całkowita liczba transkryptów (RNA) jest większa dzięki istnieniu wielu ncRNA (niekodujących RNA), alternatywnemu składaniu eksonów i innym zjawiskom.
- Jedynie niewielka część genomu — rzędu 1–2% — bezpośrednio koduje sekwencje białek. Reszta to introny, sekwencje regulatorowe, powtórzenia i elementy niekodujące, które mogą mieć funkcję strukturalną lub regulacyjną.
- Pojawiają się liczne elementy powtarzalne i ruchome (np. LINE, SINE, transpozony), które razem stanowią znaczną część genomu (blisko 40–50%).
Regulacja i organizacja funkcjonalna
Funkcja genów zależy od złożonego systemu regulacji: promotory, enhancery, czynniki transkrypcyjne, modyfikacje histonów i metylacja DNA wpływają na to, które geny są włączone w danym typie komórki i w danym czasie. Trójwymiarowa organizacja chromatyny (np. domeny TAD — topologically associating domains) umożliwia kontakt między odległymi elementami regulatorowymi a genami, co ma kluczowe znaczenie dla prawidłowej ekspresji genów.
Human Genome Project (HGP) — sekwencja referencyjna
W ramach projektu Human Genome Project opracowano sekwencję referencyjną, która jest wykorzystywana na całym świecie w biologii i medycynie. Nature opublikowało raport z projektu finansowanego ze środków publicznych, a Science opublikowało pracę firmy Celera. Prace te opisywały, w jaki sposób powstał projekt sekwencji i przedstawiały jej analizę.
HGP rozpoczął się na początku lat 90. XX w.; pierwszy roboczy szkic referencyjny genomu człowieka ogłoszono w 2001 r. (dalsze poprawki i uzupełnienia doprowadziły w latach 2003 i 2005 do wersji roboczych obejmujących do ≈92% sekwencji). Publiczne i prywatne podejścia nieco się różniły — publiczny konsorcjum stosowało mapowanie klonów i sekwencjonowanie, podczas gdy firma Celera wykorzystała metodę shotgun. Efektem prac HGP są m.in. znormalizowane odniesienia (kolejne wersje referencyjnych genomów, np. GRCh37, GRCh38), które służą jako „mapa” dla badań genetycznych i medycznych.
W kolejnych dekadach społeczność naukowa uzupełniała luki: istotnym osiągnięciem jest praca konsorcjum Telomere-to-Telomere (T2T), które w 2022 r. przedstawiło praktycznie kompletną sekwencję ludzkiego genomu (T2T-CHM13), wypełniając wiele wcześniej nieodczytanych regionów, takich jak centromery i długie powtórzenia.
Równolegle HGP zainicjował program ELSI (ethical, legal and social implications) — badania i dyskusje nad konsekwencjami społecznymi sekwencjonowania genomu, prywatnością, dostępem do danych oraz możliwą dyskryminacją genetyczną.
Projekt ENCODE — co to bada i dlaczego jest ważny
Najnowszy projekt ENCODE (Encyclopedia of DNA Elements) ma na celu zidentyfikowanie i zmapowanie elementów funkcjonalnych w ludzkim genomie — nie tylko genów kodujących białka, lecz też regulatorów, miejsc wiązania czynników transkrypcyjnych, modyfikacji chromatyny, transkryptów niekodujących i innych „elementów” wykazujących aktywność biochemiczną.
ENCODE wykorzystuje metody wysokoprzepustowe, m.in.:
- RNA-seq do wykrywania transkryptów (również niedawno odkrytych długich ncRNA i krótkich RNA),
- ChIP-seq do mapowania miejsc wiązania białek i modyfikacji histonów,
- DNase-seq / ATAC-seq do identyfikacji regionów otwartej chromatyny (potencjalnie aktywnych regulatorowo),
- techniki 3D (np. Hi-C) do badania przestrzennej organizacji genomu.
Jednym z najbardziej dyskutowanych wyników pierwszych odsłon ENCODE (2012) była deklaracja, że znacząca część genomu wykazuje "aktywność biochemiczną" — co media przedstawiły jako stwierdzenie, że 80% genomu ma funkcję. To wywołało debatę naukową: krytycy zwracali uwagę, że aktywność biochemiczna (np. chwilowe wiązanie białka lub jednorazowy transkrypt) nie musi oznaczać biologicznie istotnej, wyewoluowanej funkcji. Późniejsze analizy i kolejne wydania ENCODE lepiej rozróżniły pojęcia „biochemiczna aktywność” od „funkcji biologicznej” rozumianej przez selekcję ewolucyjną.
Zastosowania praktyczne i dalsze kierunki
Mapa referencyjna genomu i zasoby tworzone przez projekty typu ENCODE mają wiele praktycznych zastosowań:
- interpretacja wariantów związanych z chorobami — wiele wariantów wykrywanych w badaniach GWAS znajduje się nie w genach, lecz w regionach regulatorowych; mapy ENCODE pomagają określić, które warianty mogą wpływać na ekspresję genów,
- onkologia — sekwencjonowanie nowotworów i analiza zmian regulacyjnych pomagają odnaleźć kierunki terapii celowanej,
- medycyna spersonalizowana i farmakogenomika — lepsze zrozumienie funkcji genów i regulatorów umożliwia dostosowanie terapii do pacjenta,
- biotechnologia i inżynieria genetyczna — znajomość sekwencji i regulatorów ułatwia projektowanie konstrukcji genetycznych i terapii opartych na CRISPR.
Wyzwania i etyka
Mimo postępów wiele zagadnień pozostaje otwartych: pełne zrozumienie funkcji większości regionów genomu, interpretacja wariantów rzadkich, znaczenie epigenetyki i interakcji gen‑środowisko. Równocześnie rozwój technologii wymusza stałą dyskusję o ochronie prywatności danych genetycznych, odpowiedzialnym udostępnianiu wyników oraz zapobieganiu dyskryminacji na tle genetycznym.
Podsumowując: sekwencja ludzkiego genomu oraz badania takie jak ENCODE przekształciły biologię i medycynę, dostarczając ramy do zrozumienia, jak zapis genetyczny jest wykorzystywany i regulowany w komórkach. Praca nad pełnym zrozumieniem genomu trwa nadal — kolejne technologie i analizy danych wciąż odsłaniają nowe poziomy złożoności.
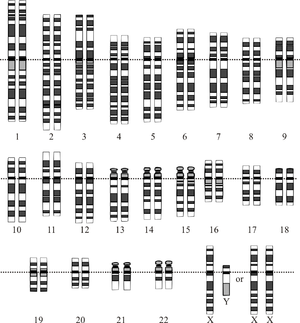
Graficzne przedstawienie wyidealizowanego kariotypu człowieka, ukazujące organizację genomu w chromosomy. Rysunek przedstawia zarówno żeńską (XX), jak i męską (XY) wersję 23. pary chromosomów.
DNA i białka
Ludzki genom zawiera nieco ponad 20 000 genów kodujących białka, czyli znacznie mniej niż się spodziewano. W rzeczywistości tylko około 1,5% genomu koduje białka, podczas gdy reszta składa się z genów niekodującego RNA, sekwencji regulatorowych i intronów.
Jednakże pojedynczy gen może produkować wiele różnych białek poprzez splicing RNA. Jeden konkretny gen Drosophila (DSCAM) może być alternatywnie splicowany do 38 000 różnych mRNA. Każdy mRNA koduje inny łańcuch peptydowy. Dlatego liczba produkowanych białek znacznie przewyższa liczbę kodujących je genów.
Biorąc pod uwagę splicing RNA i zmiany po translacji RNA, całkowita liczba unikalnych ludzkich białek może być liczona w milionach.
Pomysł, że większość DNA to bezużyteczne "śmieci" jest błędny. Co najmniej 80% genomu ma określone funkcje.
Różnice między ludźmi a szympansami
Zwierzęciem żyjącym obecnie, które jest najbliżej człowieka, jest szympans. 98,4% DNA jest takie samo u ludzi i szympansów. Dotyczy to jednak tylko polimorfizmu pojedynczych nukleotydów, czyli zmian tylko w pojedynczych parach zasad. Pełny obraz jest raczej inny.
Szkic sekwencji wspólnego genomu szympansa został opublikowany w 2005 roku. Wykazał on, że regiony, które są na tyle podobne, że można je dopasować do siebie, stanowią 2400 milionów z 3164,7 milionów zasad ludzkiego genomu, czyli 75,8% genomu.
Te 75,8% ludzkiego genomu różni się od genomu szympansa o 1,23% polimorfizmami pojedynczych nukleotydów (SNPs - zmiany pojedynczych "liter" DNA w genomie). Inny rodzaj różnic, zwany "indelami" (insercje/delecje), stanowi kolejne ~3% różnic między sekwencjami, które można ze sobą porównać. Dodatkowo, różnice w liczbie kopii dużych segmentów (> 20 kb) podobnej sekwencji DNA dostarczają kolejnych 2,7% różnic między dwoma gatunkami. Stąd całkowite podobieństwo genomów może wynosić nawet około 70%.
Powiązane strony
Pytania i odpowiedzi
P: Gdzie przechowywany jest ludzki genom?
O: Ludzki genom jest przechowywany na 23 parach chromosomów w jądrze komórkowym i w małym mitochondrialnym DNA.
P: Co obecnie wiadomo na temat sekwencji DNA na naszych chromosomach?
O: Obecnie wiadomo bardzo dużo na temat sekwencji DNA na naszych chromosomach.
P: Czym jest Human Genome Project?
O: Human Genome Project (HGP) to projekt, w ramach którego opracowano sekwencję referencyjną ludzkiego genomu.
P: Jaki procent sekwencji został wypełniony zgodnie z poprawionymi wersjami roboczymi?
O: Ulepszone wersje ogłoszone w 2003 i 2005 roku wypełniły ≈92% sekwencji.
P: Jaki jest najnowszy projekt badający sposób kontroli genów?
O: Najnowszy projekt, ENCODE, bada sposób, w jaki kontrolowane są geny.
P: Chociaż sekwencja ludzkiego genomu została w pełni określona, czy jest ona w pełni zrozumiała?
O: Nie, sekwencja ludzkiego genomu nie została jeszcze w pełni poznana.
P: Co niekodujące DNA robi w genomie?
O: Niekodujące DNA w genomie robi ważne rzeczy, takie jak regulacja ekspresji genów, organizacja chromosomów i sygnały kontrolujące dziedziczenie epigenetyczne.
Przeszukaj encyklopedię