Ewolucja ukierunkowana — definicja i zastosowania w inżynierii białek
Ewolucja ukierunkowana — jak inżynieria białek tworzy ulepszone enzymy: metody in vivo i in vitro, zastosowania w medycynie i przemyśle.
Ewolucja ukierunkowana (DE) to praktyczna metoda stosowana do otrzymywania ulepszonych enzymów i innych białek do zastosowań przemysłowych, diagnostycznych i medycznych. Polega na wielokrotnym wprowadzaniu zmienności genetycznej w sekwencji kodującej białko oraz na wybieraniu wariantów o pożądanych właściwościach, a następnie wykorzystaniu ich jako punktu wyjścia do kolejnych rund optymalizacji.
Podstawowa idea i przebieg procesu
Metoda ta jest formą inżynieria proteinowa, która naśladuje selekcję naturalną. Podstawową ideą jest przeprowadzenie genu przez powtarzające się rundy mutacji w celu stworzenia biblioteki wariantów. Z biblioteki wybiera się lub przesiewa warianty o pożądanej funkcji; zwycięskie sekwencje stanowią następnie szablon dla kolejnej rundy mutagenezy i selekcji.
Główne strategie: in vivo i in vitro
DE można prowadzić in vivo (w żywych komórkach takich jak bakterii lub drożdży) lub in vitro (w systemach wolnych w roztworze lub w sztucznych mikrokropelek). Każde podejście ma zalety i ograniczenia.
W ewolucji in vivo każda komórka (najczęściej bakterie lub drożdże) jest przekształcana za pomocą plazmidu zawierającego innego członka biblioteki wariantów. Pomiędzy komórkami różni się zwykle tylko gen interesujący, przy czym wszystkie inne elementy genetyczne i warunki są takie same. Komórki wyrażają białko albo w swojej cytoplazmie, albo na powierzchni, gdzie jego funkcję można badać w kontekście komórkowym. Ten format jest szczególnie użyteczny, gdy celem jest uzyskanie białka działającego w żywym organizmie (np. terapeutycznego enzymu, transportera lub czynnika immunologicznego).
W eksperymentach in vitro DE wykorzystuje tłumaczenie i transkrypcję in vitro do produkcji białek lub RNA wolnych w roztworze lub wewnątrz sztucznych mikrokropelek. Pozwala to na silniejszą kontrolę warunków (np. temperatury, stężenia soli, użycia rozpuszczalników) oraz na ekspresję białek toksycznych dla komórek. Ponadto eksperymenty in vitro umożliwiają generowanie znacznie większych bibliotek (do 1015 wariantów), ponieważ DNA nie musi być wprowadzane do komórek, co często stanowi ograniczenie przy metodach in vivo.
Tworzenie różnorodności (mutageneza)
- Mutageneza losowa: np. error-prone PCR — wprowadzanie błędów podczas amplifikacji, by uzyskać szeroką gamę punktowych mutacji.
- Rekombinacja: techniki takie jak DNA shuffling (mieszanie fragmentów z kilku rodziców) pozwalają łączyć korzystne mutacje z różnych molekuł.
- Site-saturation / ukierunkowana mutageneza: mutacje ograniczone do konkretnych pozycji (np. miejsca aktywnego) — przydatne w podejściu pół-racjonalnym.
- Metody in vitro (np. systemy display czy linkowanie fenotyp-gen) często łączone z bibliotekami syntetycznymi o zaprojektowanej różnorodności.
Selekcja i przesiew — jak znaleźć „zwycięzców”
Wybieranie pożądanych cech można przeprowadzać dwiema głównymi drogami:
- Selekcja — przetrwanie lub wzrost zależne od funkcji białka (np. enzym przywracający metabolizm), co pozwala na szybkie wybranie aktywnych wariantów w dużych bibliotekach.
- Przesiew (screening) — mierzenie aktywności poszczególnych wariantów za pomocą testów biochemicznych, fluorescencyjnych, chromatograficznych itp.; konieczny przy cechach ilościowych i przy mniejszych bibliotekach.
Techniki display (np. phage display, yeast display), metody oparte na rybosomie, mRNA display czy nowoczesne systemy mikrodropletów i mikrofuidyki umożliwiają bardzo wysokoprzepustowe badania funkcji i izolowanie najlepszych kandydatów.
Nowoczesne podejścia i automatyzacja
W ostatnich latach rozwinięto metody ciągłej ewolucji (np. PACE — phage-assisted continuous evolution), które pozwalają na szybkie przeprowadzanie wielu rund mutacji i selekcji bez ręcznej interwencji. Połączenie DE z sekwencjonowaniem wysokoprzepustowym i analizą bioinformatyczną umożliwia mapowanie krajobrazów adaptacyjnych, identyfikację epistaz i przewidywanie skutków kombinacji mutacji.
Zastosowania
- Optymalizacja enzymów przemysłowych (stabilność, aktywność w ekstremalnych warunkach, odporność na rozpuszczalniki).
- Ulepszanie leków białkowych i przeciwciał (większa powinowactwo, stabilność, mniejsze immunogenność).
- Projektowanie biosensorów i katalizatorów do syntezy chemicznej.
- Rozwój nowych terapii genowych, enzymów do terapii enzymatycznych i szczepionek.
- Optymalizacja ścieżek metabolicznych w mikrobach przemysłowych.
Wyzwania i ograniczenia
Chociaż DE jest potężną metodą, istnieją ograniczenia i pułapki:
- Skala biblioteki vs. zdolność przesiewu — ogromne biblioteki wymagają odpowiednio wydajnych metod selekcji/przesiewu.
- Trade-offy — zwiększenie jednej cechy (np. aktywności) może obniżyć inną (np. stabilność), dlatego często stosuje się strategie równoważenia właściwości.
- Epistaza — efekt kombinacji mutacji może być nieprzewidywalny; nie wszystkie korzystne mutacje sumują się liniowo.
- Bezpieczeństwo biologiczne i etyka — modyfikowanie funkcji biologicznych wymaga rygorystycznej oceny ryzyka, kontroli oraz zgodności z regulacjami.
Łączenie z projektowaniem racjonalnym
W praktyce często łączy się DE z podejściami racjonalnymi lub pół-racjonalnymi: analiza struktury białka, modelowanie komputerowe i mutageneza skierowana pozwalają skoncentrować bibliotekę na najbardziej obiecujących regionach sekwencji, co zwiększa efektywność procesu.
Podsumowanie
Ewolucja ukierunkowana to uniwersalne narzędzie inżynierii białek, które dzięki kombinacji wprowadzania zmienności i ukierunkowanej selekcji pozwala uzyskać białka o ulepszonych funkcjach. Wybór pomiędzy in vivo a in vitro, metoda mutagenezy i sposób selekcji zależą od celu projektu, dostępnych zasobów i ograniczeń bezpieczeństwa. Dzięki postępom w mikrofuidyce, ciągłej ewolucji i sekwencjonowaniu, DE staje się coraz szybsza i bardziej precyzyjna, otwierając nowe możliwości w biotechnologii i medycynie.
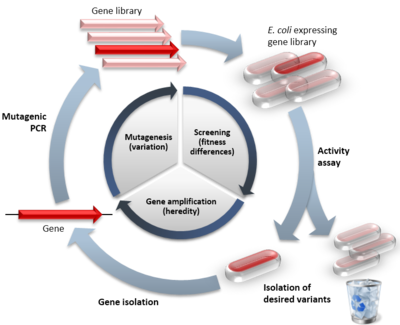
Przykład ukierunkowanej ewolucji w porównaniu z ewolucją naturalną. Cykl wewnętrzny pokazuje trzy etapy cyklu ewolucji ukierunkowanej z naśladowaniem procesu naturalnego w nawiasach. Okrąg zewnętrzny pokazuje kroki w typowym eksperymencie. Czerwone symbole oznaczają warianty funkcjonalne, blade - warianty o zredukowanej funkcji.
Zapewnienie dziedziczności
Gdy białka funkcjonalne zostały wyizolowane, konieczne jest, aby ich geny były również ich genami, dlatego wymagane jest połączenie genotyp-fenotyp.
Może to być kowalencyjne, gdzie gen mRNA jest połączony z białkiem na końcu tłumaczenia przez puromycynę.
Alternatywnie, białko i jego gen mogą być trzymane razem, lub w kroplach emulsji. Wyizolowane sekwencje genów są następnie rozmnażane za pomocą PCR lub przez przekształcone bakterie gospodarza. Albo pojedyncza najlepsza sekwencja, albo pula sekwencji może być użyta jako wzorzec dla następnej rundy mutagenezy. Powtarzające się cykle zróżnicowania-selekcji-amplifikacji powodują, że warianty enzymatyczne są dostosowane do procesu selekcji.

Wyrażone białko może być połączone kowalencyjnie z jego genem (jak w mRNA), po lewej stronie, lub umieszczone w tej samej komorze z nim, po prawej stronie. Tak czy inaczej, gen, który koduje białko, jest izolowany
Nagroda przyznana
Amerykański inżynier FrancesArnold otrzymał nagrodę Millennium Technology Prize za pionierską, ukierunkowaną ewolucję.
Pytania i odpowiedzi
P: Co to jest ewolucja ukierunkowana?
O: Ewolucja kierowana (DE) jest metodą stosowaną do produkcji enzymów dla celów przemysłowych lub medycznych. Jest to forma inżynierii białek, która naśladuje dobór naturalny.
P: Jak działa ewolucja kierowana?
O: Ewolucja ukierunkowana polega na poddawaniu genu wielokrotnym mutacjom, tworząc bibliotekę wariantów. Selekcja wyodrębnia geny o pożądanej funkcji, które są następnie wykorzystywane jako szablony do następnej rundy.
P: Gdzie można przeprowadzić ewolucję ukierunkowaną?
O: Ewolucję ukierunkowaną można przeprowadzać in vivo (w żywych komórkach bakterii lub drożdży) lub in vitro (w roztworze lub mikrokroplach).
P: Jakie są zalety prowadzenia ewolucji kierowanej in vivo?
O: Zaletą ewolucji kierowanej in vivo jest możliwość wyboru właściwości w środowisku komórkowym, co jest przydatne, gdy wyewoluowane białko lub RNA ma być wykorzystane w organizmach żywych.
P: Jakie są zalety prowadzenia ukierunkowanej ewolucji in vitro?
O: Zaletą ewolucji kierowanej w warunkach in vitro jest to, że pozwala ona na stworzenie większej liczby warunków (np. temperatura, rozpuszczalniki) i umożliwia ekspresję białek, które byłyby toksyczne dla komórek. Ponadto można generować znacznie większe biblioteki, ponieważ DNA nie musi być wprowadzane do komórek.
P: Co ogranicza to, co można zrobić podczas eksperymentu in vitro?
O: Limit wielkościowy tego, co można zrobić podczas eksperymentu in vitro, jest często określony przez to, ile DNA trzeba wprowadzić do komórek.
Przeszukaj encyklopedię